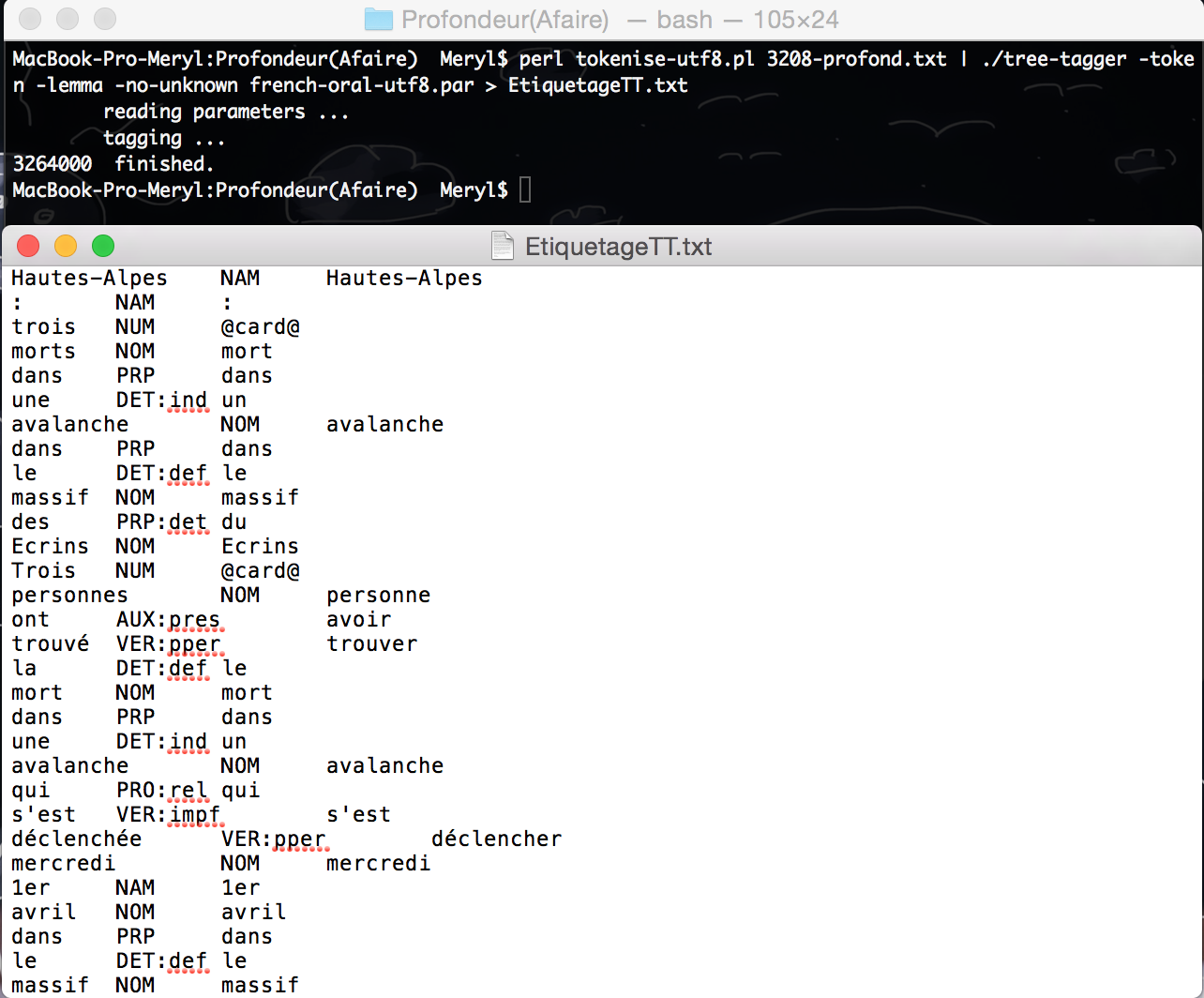
La BAO2 est l'étape qui nous permet d'étiqueter morphosyntaxiquement nos fichiers obtenus dans la BAO1.
Nous avons procédé à l'étiquetage de deux manières différentes :
1° CORDIAL
Fiche technique de Cordial:
NE TRAITE QUE LA SURFACE DES FICHIERS TEXTES
ENTREE : fichiers textes de surface
SORTIE : fichiers textes de surface étiquetés
NE TRAITE QUE LA SURFACE DES FICHIERS TEXTES
ENTREE : fichiers textes de surface
SORTIE : fichiers textes de surface étiquetés
Télécharger les fichiers obtenus
2° TREETAGGER
Fiche technique du script :
TRAITEMENT DE LA SURFACE
ENTREE : fichiers txt de surface extraits en BAO1
SORTIE : fichiers xml contenant le texte et son étiquetage
TRAITEMENT DE LA SURFACE
ENTREE : fichiers txt de surface extraits en BAO1
SORTIE : fichiers xml contenant le texte et son étiquetage
#/usr/bin/perl
#-----------------------------------------------------------
# le premier argument est le nom du repertoire contenant les fichiers XML a etiqueter
my $rep="$ARGV[0]";
# le second argument est le nom de la rubrique
my $rubrique = "$ARGV[1]";
my %dico;
# on s'assure que le nom du répertoire ne se termine pas par un "/"
$rep=~ s/[\/]$//;
#----------------------------------------
# on initialise une variable contenant le flux de sortie
my $sortieTXT="$rubrique.txt";
if (!open (FILEOUT,">$sortieTXT")) { die "Pb a l'ouverture du fichier $sortieTXT"};
close(FILEOUT);
my $sortieXML="$rubrique.xml";
if (!open (FILEOUT,">$sortieXML")) { die "Pb a l'ouverture du fichier $sortieXML"};
print FILEOUT "\n";
print FILEOUT "\n";
print FILEOUT "BOTHUA \n";
close (FILEOUT);
#----------------------------------------
&parcoursarborescencefichiers($rep); # on lance la récursion.... et elle se terminera après examen de toute l'arborescence
#----------------------------------------
open (FILEOUT,">>:encoding(utf-8)", $sortieXML);
print FILEOUT " \n";
close(FILEOUT);
exit;
#----------------------------------------------
sub parcoursarborescencefichiers {
# variable spéciale @_ (qui est donc un tableau) : shift recupere l'element de _@ qui est le premier argument
my $path = shift(@_);
# ouvrir du dossier qui est celui entré en argument
opendir(DIR, $path) or die "can't open $path: $!\n";
#créer une liste qui contient les éléments du répertoire
my @files = readdir(DIR);
closedir(DIR);
# pour chaque fichier ou dossier dans l'arborescence parcourue
foreach my $file (@files) {
next if $file =~ /^\.\.?$/;
next if $file =~ /^\._/;
next if $file =~ /^fil/;
$file = $path."/".$file;
# si on tombe sur un repertoire, on va le parcourir et donc relancer la fonction parcoursarborescencefichiers
if (-d $file) {
print " ==> ",$file,"\n";
&parcoursarborescencefichiers($file);
print " ==> ",$file,"\n";
}
# si on tombe sur fichier, on va le traiter
if (-f $file) {
if ($file =~ /$rubrique.+\.xml$/) {
print "<",$i++,"> ==> ",$file,"\n";
$codage = "utf-8";
open (FIC, "<:encoding($codage)", $file);
# concaténation des informations récoltées dans les fichiers instanciés en début de programme
open (OUTTXT, ">>:encoding($codage)", "$rubrique.txt");
open (OUTXML, ">>:encoding($codage)", "$rubrique.xml");
#ramener tout le flux textuel de FIC sur une seule ligne
my $texte="";
while (my $ligne = ) {
chomp $ligne;
$ligne =~ s/\r//g;
$texte = $texte . $ligne;
}
close FIC;
$texte =~ s/>\s+([^<]+?)<\/title><link>(?:[^<]+?)<\/link><description>([^<]+?)<\/description>/g) {
my $titre = $1;
my $description = $2;
$titre=~s/<.+?>//g;
$titre.=".";
$titre=~s/\?\.$/\?/;
$description=~s/<.+?>//g;
if (!(exists $dico{$titre})) {
$dico{$titre} = 1;
print OUTTXT "$file \n";
print OUTTXT "$titre\n";
print OUTTXT "$description\n\n";
my ($titretag,$descriptiontag) = &etiquetage($titre,$description);
print OUTXML "<item file=\"$file\"><title>$titretag $descriptiontag \n";
}
}
close OUTTXT;
close OUTXML;
# fin de traitement du fichier
}
}
}
}
sub etiquetage {
my ($t,$d)= @_;
open(TMP,">:encoding(utf8)","titre.txt");
print TMP $t;
close TMP;
# system permet de lancer une commande bash dans le terminal
system("perl tokenise-utf8.pl titre.txt | ./tree-tagger -token -lemma -no-unknown french-oral-utf8.par > titre_tag.txt");
system("perl treetagger2xml-utf8.pl titre_tag.txt utf8"); #résultat est contenu dans titre_tag.txt.xml
open(TMP2, "<:encoding(utf8)","titre_tag.txt.xml");
my $t_tag="";
my $ligne = ;
while (my $ligne = ) {
#chomp $ligne;
$t_tag = $t_tag . $ligne;
}
close TMP2;
open(TMP,">:encoding(utf8)","description.txt");
print TMP $d;
close TMP;
system("perl tokenise-utf8.pl description.txt | ./tree-tagger -token -lemma -no-unknown french-oral-utf8.par > description_tag.txt");
system("perl treetagger2xml-utf8.pl description_tag.txt utf8"); #résultat est contenu dans titre_tag.txt.xml
open(TMP2, "<:encoding(utf8)","description_tag.txt.xml");
my $d_tag="";
my $ligne = ;
while (my $ligne = ) {
#chomp $ligne;
$d_tag = $d_tag . $ligne;
}
close TMP2;
return($t_tag,$d_tag);
}
#----------------------------------------------
Télécharger les fichiers obtenus
Fiche technique TreeTagger :
TRAITEMENT DE LA PROFONDEUR
ENTREE : fichiers txt profond extraits en BAO1
SORTIE : fichiers txt des articles étiquetés
TRAITEMENT DE LA PROFONDEUR
ENTREE : fichiers txt profond extraits en BAO1
SORTIE : fichiers txt des articles étiquetés